24년 6월 4주차 그래프 오마카세

A joint Graph and Image Convolution Network for Automatic Brain Tumor Segmentation
BrainLes 2021
Keywords
Brain tumor segmentation, 3D supervoxel clustering, Graph representation learning, CNN refinement
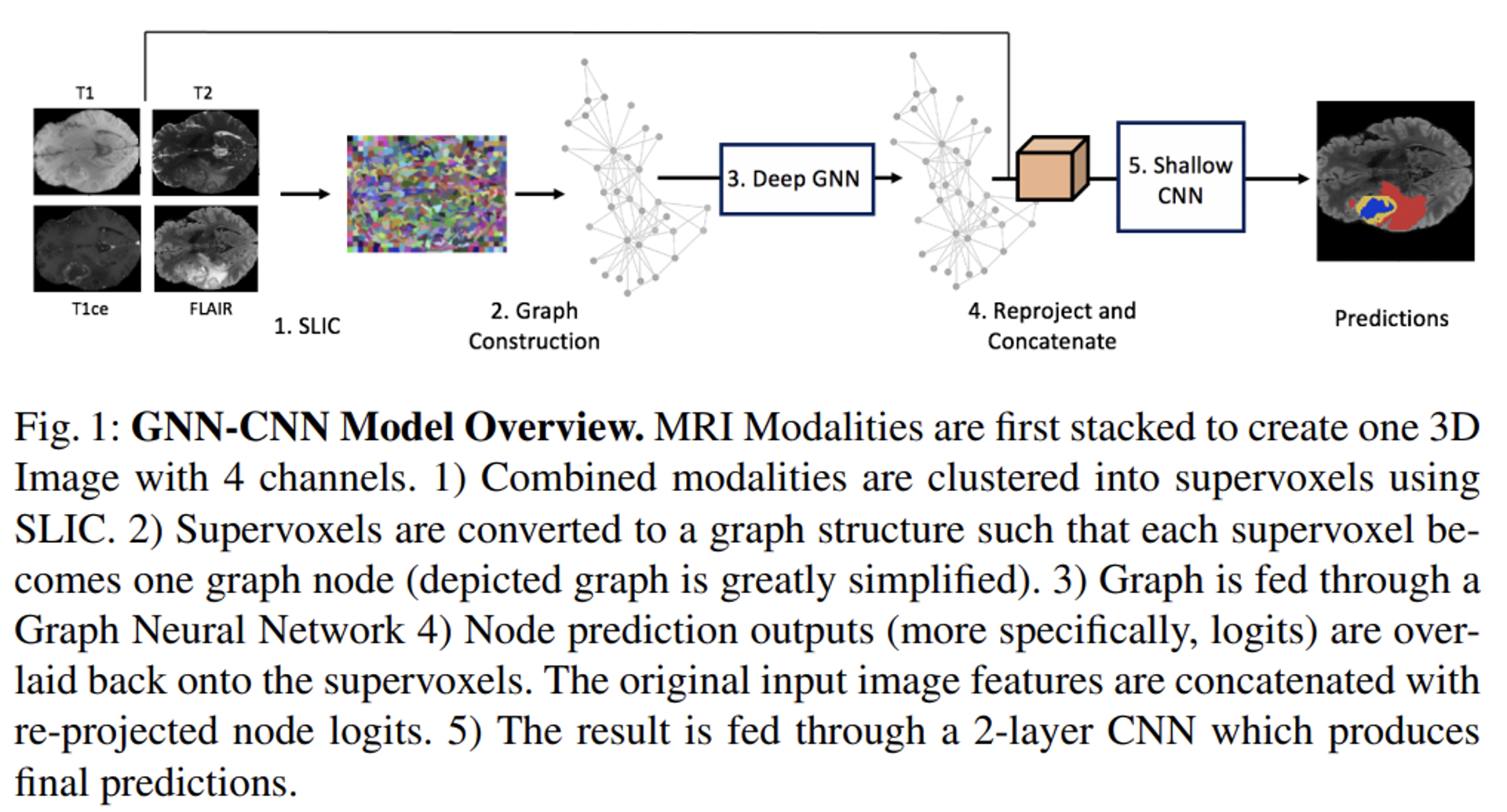
Summary
- 이번에 소개해드릴 논문은 메디컬 이미징 뇌종양 분할 챌린지 (Brain tumor segmentation challenge, BRAT) 2021에 투고된 것으로, 저번에 소개해드린 Supervoxel-based graph representation learning의 성능적 한계를 해결하기 위한 방법을 제안합니다.
- 간략하게, 그래프 도메인에서 이미지 처리를 위한 supervoxel 방법의 효과는 클러스터링된 이미지 상에 표현된 입력 그래프의 크기를 줄이면서도 각 노드가 이미지의 중요 특징정보를 포함하게 되어 효과적인 학습과 좋은 성능을 얻어낼 수 있다는 점입니다.
- 하지만, 다음 설명은 supervoxel 알고리즘의 최적 결과로 얻어진 이미지와 그래프 표현 알고리즘의 조화가 잘 맞아떨어졌을 때 해당하는 가정입니다. 즉, 이러한 효과를 손쉽게 얻어낼 수 없고, 더욱이나 알고리즘의 기반이 대부분 하이퍼파라미터이기 때문에 직접 최적의 파라미터를 찾아 선택해주어야 한다는 단점이 존재합니다.
- 주어진 이미지 데이터셋 마다 요구되는 최적의 파라미터 값들이 다를 수 있기 때문에, 모든 이미지 상에 이러한 파라미터 값을 찾아내는 것은 불가능합니다.
- 대체적으로 hyperparam searching 기법을 활용하고 있으나, 여전히 그 성능은 미미한 편입니다.
- 이러한 큰 한계를 해결하기 위하여, 다음 저자들은 Refinement를 추가하여 학습된 그래프 정보들이 주어진 도메인 상에 더욱 적합해질 수 있도록 만들고자 합니다. 다음 Refinement 단계의 베이스는 simple CNN (Convolutional Neural Network)입니다.
- 복잡한 모델을 Refinement로 사용하기엔 연산량이 크게 증가할 수 있기 때문에, 저자들은 단순 구조의 CNN 모델을 추가하였으며 이것으로도 충분히 좋은 성능 향상을 얻어낼 수 있었다고 합니다.
- 구체적으로, 이전 방법과 동일하게 주어진 입력 MRI modalities (T1, T2, T1CE, FLAIR)들을 concat하여 4채널의 MRI tensor를 만들고, 그 위에 클러스터링 알고리즘 SLIC을 통해 Supervoxel를 생성합니다. Supervoxel의 최적 파라미터를 찾아내기 위해 저자들은 클러스터링 개수 및 compactness 계수에 대하여 Grid-search 기법을 사용했다고 합니다. 다음은 클러스터링의 성능을 평가하기 위한 ASA(Achievable Segmentation Accuracy) 지표를 통해 최적화됩니다.
- 얻어진 supervoxel 위에 그래프를 형성하기 위해 일반적인 KNN 방식을 활용하지 않고, 각 복셀을 그래프 노드로 지정 후 복셀 간 유사도를 계산하여 인접 행렬을 구축하고 그를 기반으로 무가중치 엣지를 형성합니다. 여기에서 KNN 방식을 활용하지 않고 인접행렬을 구축한 이유로, 일정 반경 내 모든 불필요한 영역 간의 연결을 방지하여 연산적인 효율성을 얻어내기 위함과, 직접 구축한 인접 행렬이 그래프의 표현력을 향상시켜주기 때문입니다.
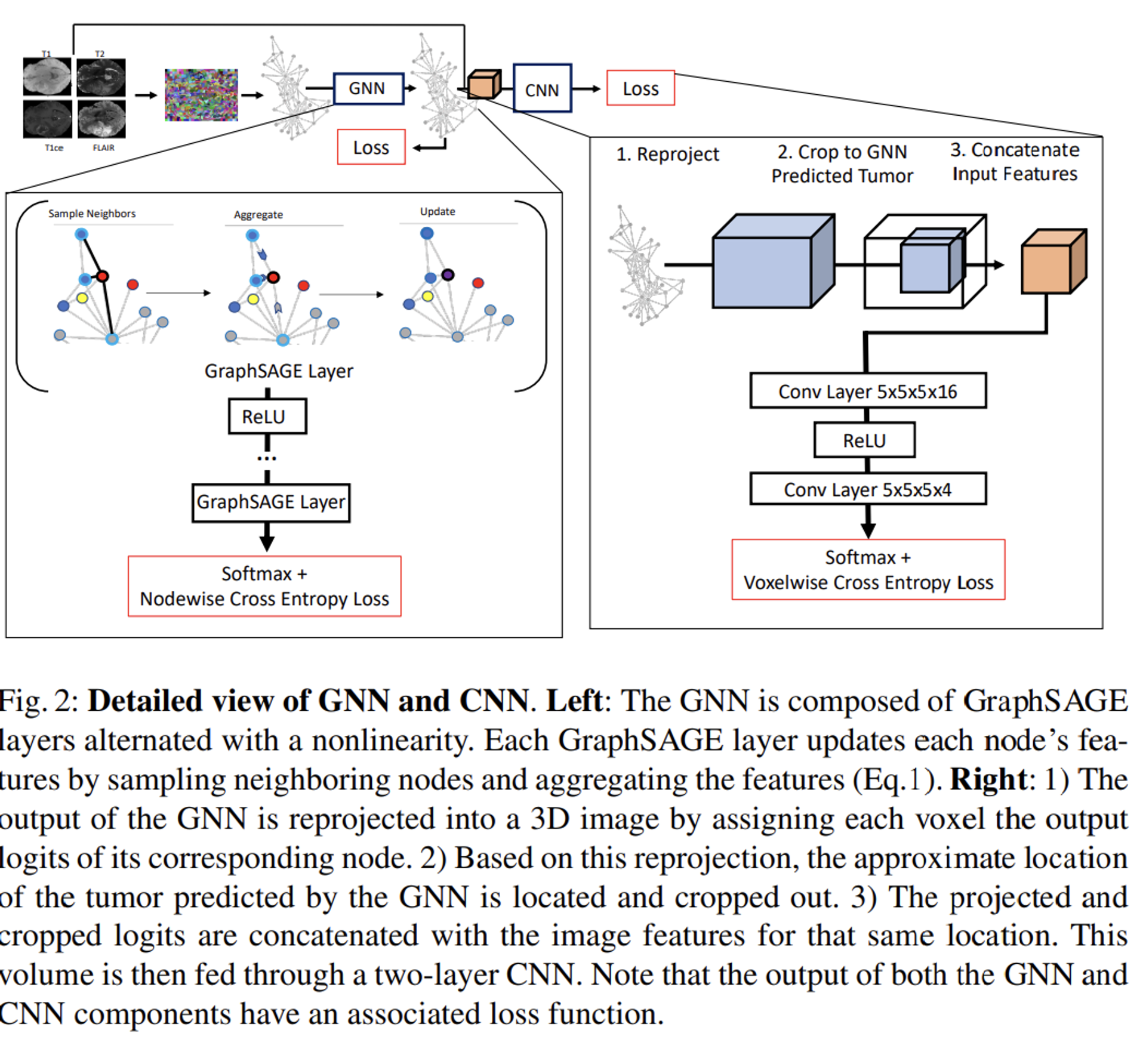
- 그리고 GraphSAGE 모델을 baseline으로 하여 해당 supervoxel graph를 학습합니다. 해당 학습은 nodewise CE loss를 통해 supervised되어지며 최종 그래프 정보가 업데이트된 결과를 출력합니다. 해당 gradient는 SAGE 모델만을 통해 backpropagate 됩니다. 구체적 모델 구조는 Fig 2의 왼쪽을 참고하시면 되겠습니다.
- 출력된 그래프를 바로 prediction에 사용하지 않고, 위에서 언급한 문제점들을 보완하기 위해, 저자들은 GNN 학습을 통해 뽑힌 tumor prediction에 대해 추가 refinement를 진행합니다. 먼저 출력 그래프를 다시 이미지 도메인으로 mapping시킨 이후, crop된 tumor prediction에 대한 feature matrix를 재구축합니다. 다음 feature matrix에 대하여 2개 레이어로 구성된 3D CNN 모델을 설계하여 이를 재학습시킵니다. 구체적인 과정은 Fig 2의 오른쪽을 참고하시면 되겠습니다.
- 이미지에서 강력한 성능을 보장할 수 있는 CNN 모델의 추가로 인해, GNN-based tumor prediction의 불확실한 성능을 정제할 수 있고, 전체 이미지가 아닌 crop된 specific region에 대하여 학습을 진행하기 때문에, Simple한 CNN 구조만으로도 성능향상의 효과를 얻어낼 수 있었습니다.
- 다음 학습은 Voxelwise CE loss를 통해 supervised되어집니다. 마찬가지로 다음 gradient는 CNN 레이어로만 backpropagate됩니다.
- 위의 Backpropagation 과정을 만족하면서 학습 시간을 단축하기 위하여 저자들은 GNN 및 CNN 모델을 동시에 학습시키지 않았다고 합니다.
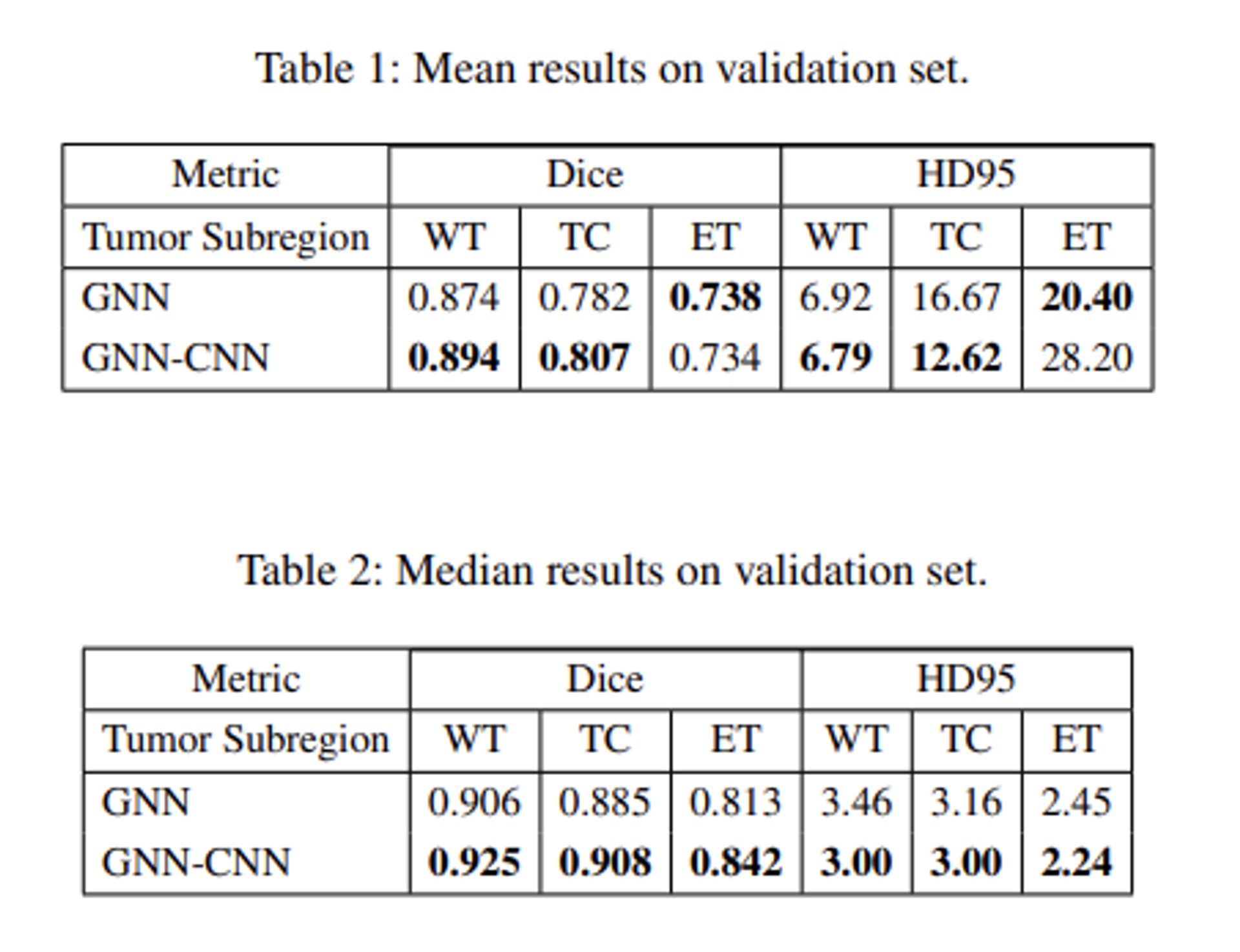
- Segmentation의 성능을 검증하기 위하여 두 가지 평가지표 (Dice, Hausdorff distacne)를 사용하였습니다.
- 실험 결과에서 단일 GNN으로 얻어낸 결과보다 CNN refinement를 추가한 제안 모델이 전체 지표에서 향상된 성능을 보여주었습니다. 즉, Refinement 과정을 통해 성공적으로 GNN의 오분류 prediction을 잘 정제하였다는 사실을 설명할 수 있습니다.
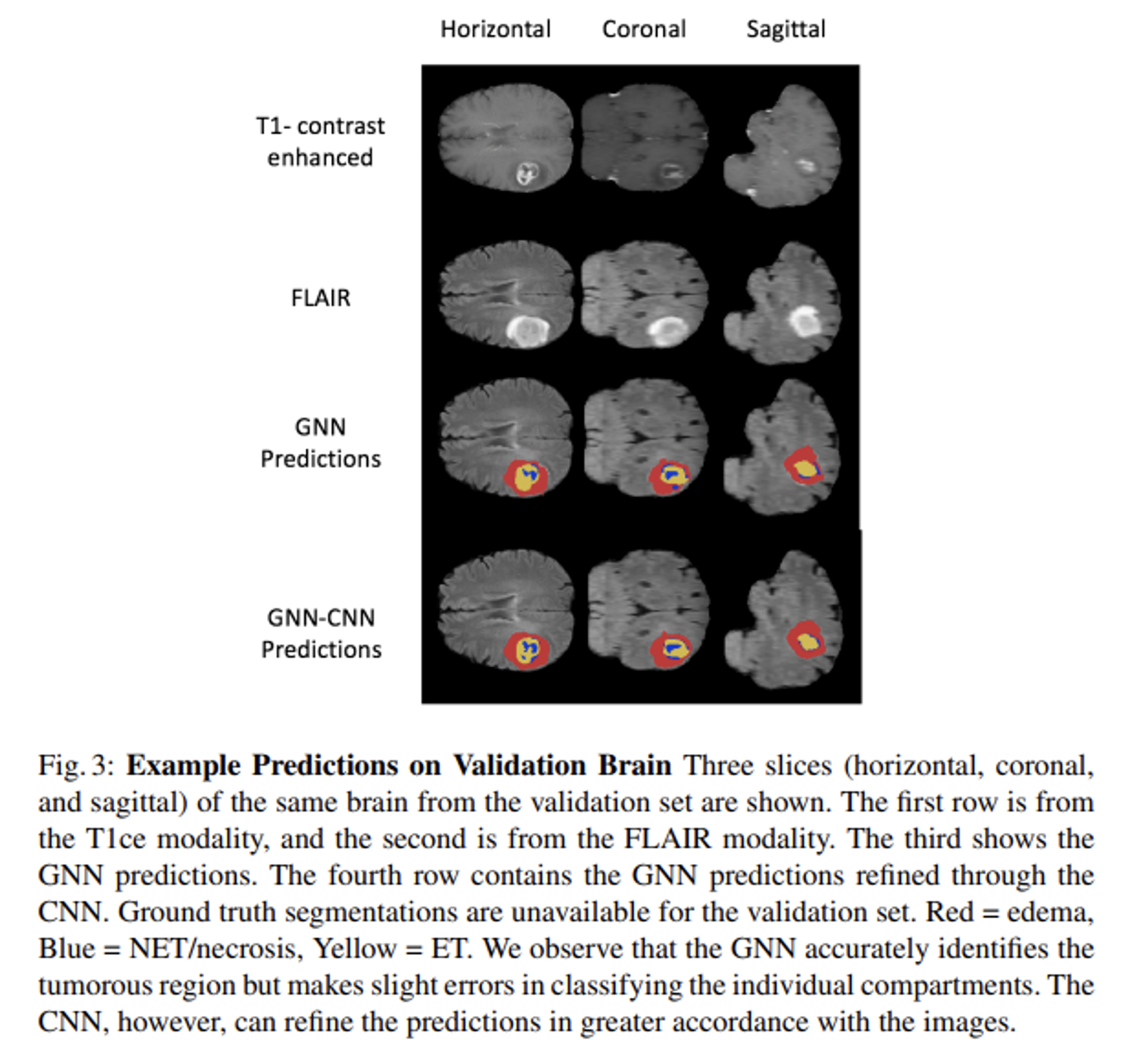
- 정성적 결과를 통해서, T1CE 및 FLAIR 모달리티로 촬영된 MRI 이미지에 보이는 종양 영역에 대한 Whole Tumor (WT)에 적합한 메커니즘으로 동작함을 알 수 있으며, CNN-refined prediction은 두 모달리티에서 종양의 형태 혹은 appearance에 대해 더욱 잘 대응할 수 있다는 사실을 알 수 있습니다.
- 해당 논문을 간략히 요약하면, 뇌종양 분할 application에서 추가 CNN refinement 과정을 통해 기존 supervoxel-based 단일 GNN 학습만으로 발생할 수 있는 한계 및 오분류 문제들을 잘 컨트롤하고 성능 향상의 효과를 얻어낼 수 있는 전체적인 방법을 잘 설명해주었습니다.
[Contact Info]
Gmail : jhbae1184@akane.waseda.jp
Twitter (X): @jhbae1184
