25년 6월 4주차 그래프 오마카세
Boltz-2 — Towards Accurate and Efficient Binding Affinity Prediction
blog link : https://boltz.bio/boltz2
official code : https://github.com/jwohlwend/boltz
- 이번 주 그래프 오마카세는 신약 AI 도메인에서의 새롭고 짚고 넘어가기 좋은 내용들을 찾아 전달해드리고자 합니다. 관심 있으신 독자 여러분들께 많은 도움이 되셨으면 좋겠습니다.
- MIT와 AI 신약개발 대표기업으로 자리잡은 Recursion이 Alphafold 3의 가장 성공적인 오픈소서 버전인 Boltz-2를 출시하였습니다. 구조(structure) 및 친화도(affinity) 예측을 통합하여 이전 Boltz-1 버전보다 훨씬 빠른 속도로 전례 없는 결합 정확도를 제공하여 신약 개발의 경계를 넓혀주었다는 평을 받고 있습니다.
- 물리 시뮬레이션보다 수십 배 빠른 속도를 갖는 Boltz-2는 추론 속도의 향상을 위해 SynflowNet을 결합하여 TYK2 단백질의 결합자를 생성하는 실험도 해당 논문을 통해 제공하고 있습니다. 또한 깃허브에 MIT 라이센스로 공개된 코드와 모델 가중치를 제공하여 추가 발전을 위한 열린 네트워크를 마련합니다.
- 해당 논문의 내용과 아키텍처에 관련한 내용을 가볍게 요약해보았습니다.
- Boltz-2는 AlphaFold 3 및 Boltz-1의 후속 모델로써, 기존의 약물 설계에 중요하지만 binding affinity 예측 능력에 있어서 기존 방법 (Free Energy Purturbation - FEP, docking) 등의 느린 동작 및 낮은 정확도의 문제 등을 해결하기 위해 방법 컨디셔닝, 거리 제약 및 멀티체인 템플릿 통합(method conditioning, distance constaints, multi-chain template integration) 등의 다양한 제어 가능성(controllabillity) 기능을 도입하였습니다.
- 작은 분자 생성모델과의 결합을 통해 TYK2 target에 대한 ABFE (absolute FEP) 시뮬레이션으로 검증된 다양하고 합성 가능한 high-affinity binder를 찾는 효과적인 워크플로우를 제공합니다.
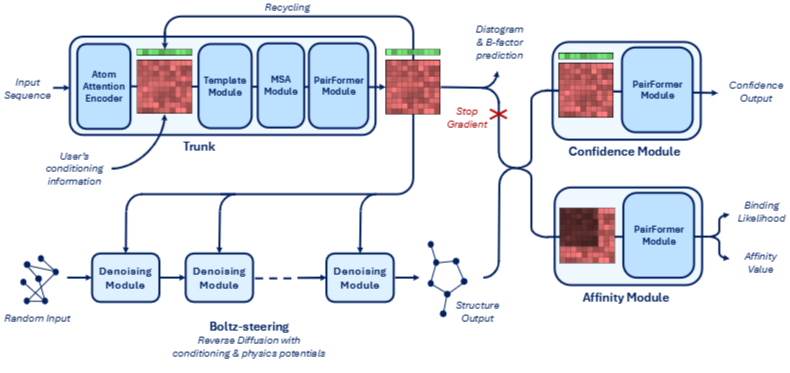
- Fig 2의 Boltz-2 모델 구조를 살펴보면 네가지 주요 구성요소로 이루어져 있음을 알 수 있습니다.
- Trunk : 모델의 핵심 부분으로, 입력 시퀀스와 사용자의 조건 정보를 처리하는 단입니다. Atom Attention Encoder (초기 입력된 복합체의 각 구성요소에 대한 기본적인 표현 인코딩), Template Module (예측하려는 구조에 대한 사전 지식을 통합), Multiple Sequence Alignment (MSA) Module (단백질 서열의 Evolutionary co-variation 및 보존된 패턴을 학습), PairFormer Module (복합체 내 모든 토큰 쌍 간의 관계와 상호작용에 대한 정보를 고밀도로 인코딩) 등의 서브 모듈을 포함하며 단백질과 리간드 상호작용에 대한표현들을 Recycling 루프를 통해 반복적으로 개선하고 생성합니다.
- Boltz-steering : 노이즈 제거 모듈로써 무작위 입력에서 시작하여 노이즈 제거 과정을 거쳐 구조 출력을 생성합니다. 여기에서 물리적 포텐셜 (physics potentials : 비현실적 오류를 범한 구조에 대한 패널티를 부여하는 함수.)을 활용하는 Reverse Diffusion(디노이징) 과정을 통해 Trunk에서 생성된 표현을 정제하는 역할을 담당합니다.
- Confidence Module : Trunk에서 출력된 표현을 입력받아 신뢰도 출력을 생성하는 모듈로써 PairFormer 모듈을 통해 예측된 구조의 신뢰도를 평가합니다. Stop gradient 표시를 통해 해당 모듈의 학습이 Trunk의 가중치 업데이트에 직접적인 영향을 주지 않도록 설계되었습니다.
- Affinity Module : 결합 가능성 및 결합 친화도의 두 가지 값을 예측하는 헤드를 포함한 리간드 내 상호작용에 초점을 맞춘 PairFormer 모듈을 활용합니다. 마찬가지로 Stop gradient 표시를 통해 해당 모듈의 학습이 Trunk의 가중치 업데이트에 직접적인 영향을 주지 않도록 설계되었습니다.
- 그로부터 구조 및 친화도 예측 태스크 모두에서 상당한 발전을 이루었으며, FEP 수준의 정확도와 효율성을 제공하여 약물 발견 워크플로우를 개선할 잠재력을 보여줍니다. 핵심 메커니즘 및 실험 논의에 대한 자세한 내용은 본 논문을 참고해보시면 좋을 것 같습니다.
Equivariance is dead, long live equivariance?
blog link : https://chaitjo.substack.com/p/transformers-vs-equivariant-networks
- 분자 시스템 상의 물리적 대칭성 (Physical symmetric)을 그래프 신경망의 inductive bias로 도입하는 연구를 진행해온 K. Joshi 선생님의 글을 가져왔습니다. 딥러닝 아키텍처의 범용적인 등가성(Equivariance)을 고려하는 측면에서 유용한 인사이트를 제공하고 있습니다.
- 해당 게시글에는 크게 2가지 측면에서 등가성의 필요성을 리뷰 및 검토합니다.
- Interatomic potentials for molecular dynamics : 모델의 용량을 제한함으로써 도움이 될 수 있는 저데이터 영역
- Generating molecular structure with diffusion : 데이터로부터 대칭성을 학습할 수 있는 고데이터 영역 (e.g. 생성 모델링)
- 회전 등가성 (Roto-translation equivariance : 입력 데이터가 회전되고 변환되더라도 일관된 기능 표현을 유지하는 모델의 능력) 및 에너지 보존 필요성 등을 언급하며, 엄격하게 등가 모델링 기법 (e.g. Equigrad) 없이 다음 두가지 성질의 필요성 및 달성 방법에 대한 개인적인 견해를 추가하였습니다. 또한 다음 주제와 관련한 몇 편의 최신 논문들도 소개되어있으니 관심이 있으시면 같이 생각해보면 좋을 것 같습니다.
[Contact Info]
Gmail: jhbae1184@akane.waseda.jp
Twitter (X): @jhbae1184