24년 7월 1주차 그래프 오마카세

Predicting isocitrate dehydrogenase mutation status in glioma using structural brain networks and graph neural networks.
MICCAI 2021
배지훈
Keyword
Brain tumor, IDH mutant Glioma prediction, Structural Brain Network, Graph representation learning, Interpretable GNN
Summary
- 이번 주 소개해드릴 논문에는 IDH mutational 'Glioma'의 돌연변이 성질을 예측하는 구조적 뇌 네트워크 구축 및 해당 그래프 표현 학습 방법을 소개해드리고자 합니다.
- 일반적으로 Glioma는 기존 종양 타입보다 뇌 전체 영역으로부터의 침투력이 매우 강한 돌연변이 악성 종양입니다. 예측하기 어려운 돌연변이적 특징 때문에 다음 종양을 정확하게 포착하는 메서드를 정의하는 것은 상당히 까다로우며 따라서 관련 의료 시스템 구축에 큰 어려움을 초래하고 있습니다.
- 입력 MRI 멀티모달 시퀀스를 활용하여 Glioma라는 악성 종양을 포착하는 복합적 뇌 네트워크를 효과적으로 구축하고 어떻게 그래프로 일반화시킬 수 있는지 보여주며, 그래프에 대한 GNN의 강력한 표현력을 바탕으로 학습된 종양 특징의 사전 정보들을 정확하게 예측할 수 있음을 설명하여 줍니다.
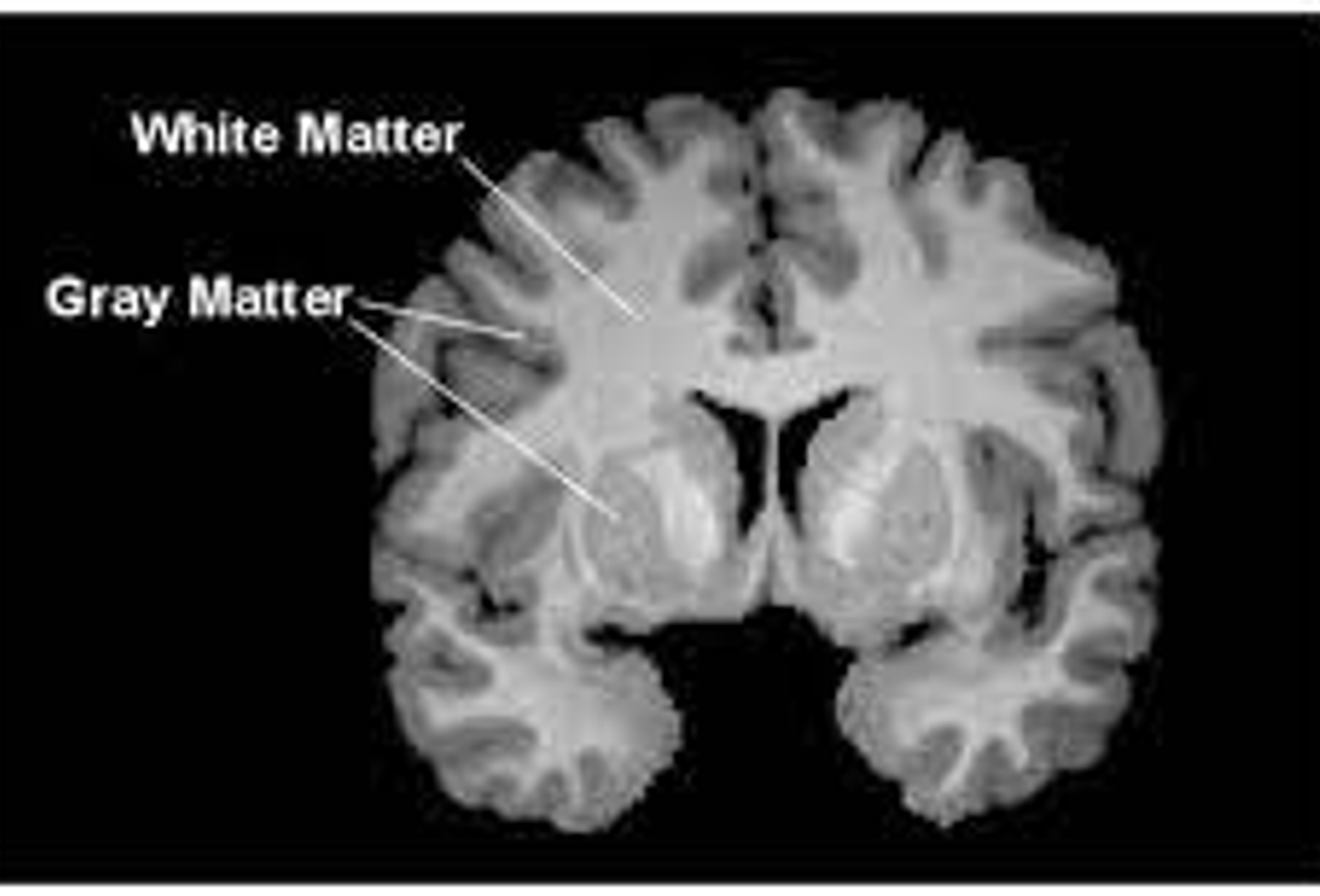
- 대뇌피질을 구성하는 회백질 (Gray Matter) 영역은 뇌 기능 사이에 더욱 명시적인 연관성을 제공합니다. 다음은 피질 및 하위피질 영역으로 구분지어 바라볼 수 있는데, 백색질 (White Matter) 영역에는 이들 사이의 축삭돌기를 포함하고 있습니다.
- 이러한 사실을 바탕으로, 구조적 뇌 네트워크 (Structural Brain Network)는 피질 및 하위피질 영역들 간 연결성 관련한 수학적 모델링 아키텍처로 바라볼 수 있습니다. 다음을 그래프로 표현하는 데 있어서, 노드를 각 영역으로, 엣지들은 백색질 부분을 연결하는 것으로 손쉽게 정의할 수 있습니다.
- Glioma 악성 세포들은 백질 (Matter) 전체의 경로를 통해 침범하여 전체적인 뇌를 공격할 수 있습니다. 다음과 관련하여 해당 논문은 그래프로 잘 표현된 구조적 뇌 네트워크 활용이 이러한 IDH mutant들의 상태를 예측하는 데 효과적인 가치를 제공할 수 있음을 설명합니다.
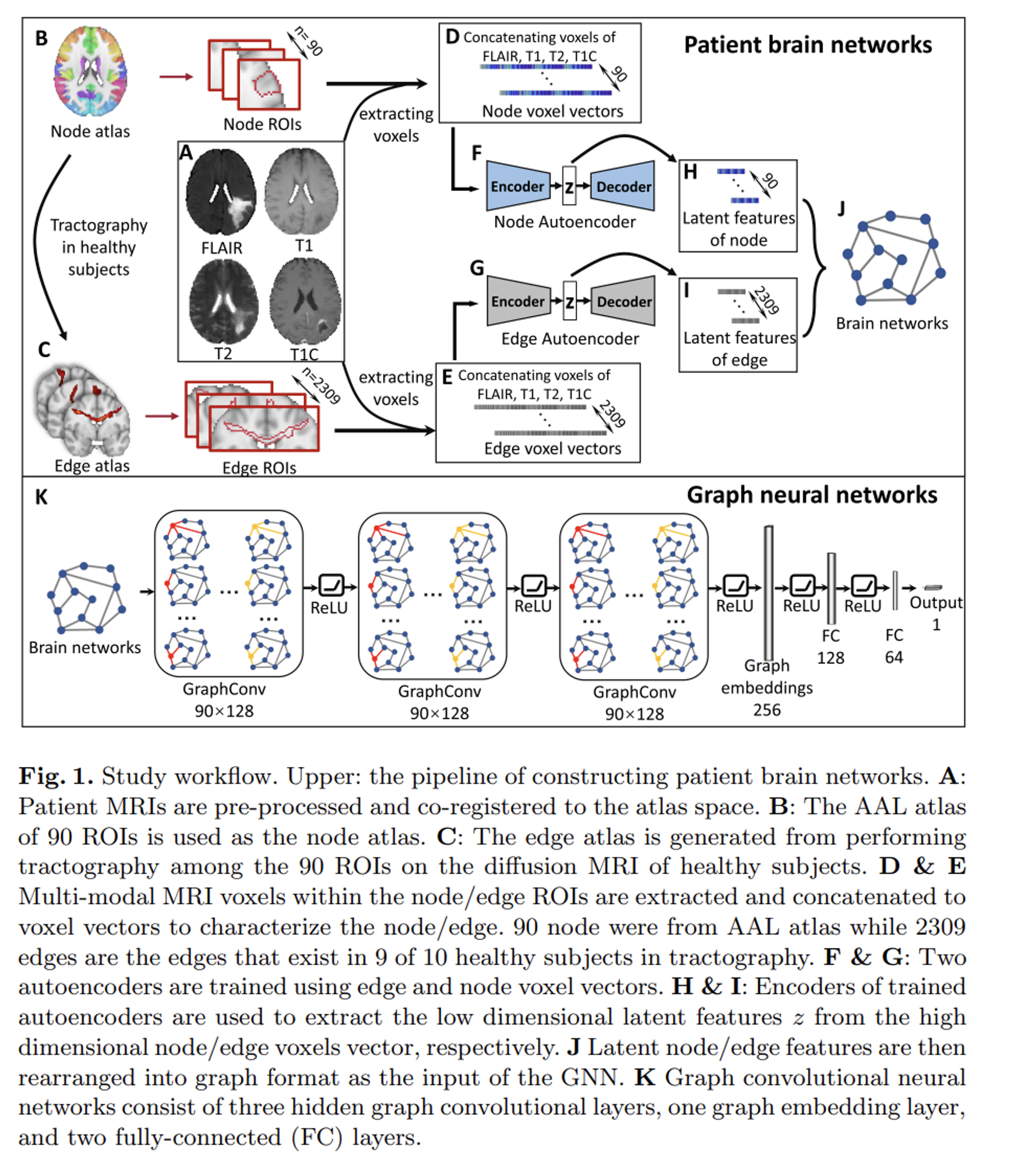
- 제안 방법론에 대하여 자세한 내용을 설명드리겠습니다. 먼저 입력 MRI 전처리 및 그래프 구성 방법은 아래와 같습니다. (Fig 1 윗부분)
- 먼저 T1, T2, FLAIR 모달리티를 post-contrast T1으로 co-register 한 이후 Voxelization 과정을 수행합니다. 다음은 DICE score를 통한 cross-validation을 통해 검증되어지며 최종적으로 출력된 복셀들을 추출합니다. (Fig 1A)
- 건강한 환자의 뇌 아틀라스 (AAL Atlas)를 템플릿으로 사용하여 90개 영역의 노드 ROI (Fig 1B의 색상부분)와 위에서 추출된 복셀들을 concatenate하여 Node voxel vectors를 생성합니다.
- Autoencoder를 활용하여 Node voxel의 latent feature을 추출합니다.
- 엣지같은 경우, AAL Atlas 상의 pairwise tractography를 적용하여 Edge ROI를 생성합니다. 각각의 ROI에 대하여 tract 밀도의 상위 5% 영역에 해당하는 복셀을 매칭시키어 binarization을 통해 최종 엣지 ROI를 출력합니다. 노드 방식과 동일하게 엣지 ROI 역시 추출 복셀들과 concatenate하여 Edge voxel vector를 생성합니다.
- 그리고 개별적인 다른 Autoencoder를 통해 latent feature을 추출합니다.
- 여기에서 두 Autoencoder는 노드 복셀 및 엣지 복셀 각각에 대하여 독립적으로 학습되어 독립적인 latent feature를 출력합니다.
- 최종적으로 출력된 Node & Edge feature를 GNN의 입력으로 사용되어 학습되어집니다. (Fig 1 아랫부분)
- 3개의 그래프 컨볼루션 레이어(GCNconv)와 2개의 FC 레이어로 구성된 GNN 네트워크를 사용합니다.
- 학습 과정에서 특별한 부분은 없으며, 데이터 불균형 문제 완화를 위해 추가적으로 Randome edge drop 기법을 활용하여 데이터를 증강시켜 활용하였으며, 학습 안정화를 위해 learning decay, early stopping 등등의 전통적 기법을 활용하였습니다.
- 실험에서 벤치마크 모델로 3D-DenseNet 및 3D-CNN 모델을 사용하였고 전체 MRI 영역 및 추출된 Tumor ROI 영역을 기준으로 한 2가지 입력 데이터에 대하여 2가지 실험방향을 설정하고 제안 모델의 성능을 비교 검증하였습니다.
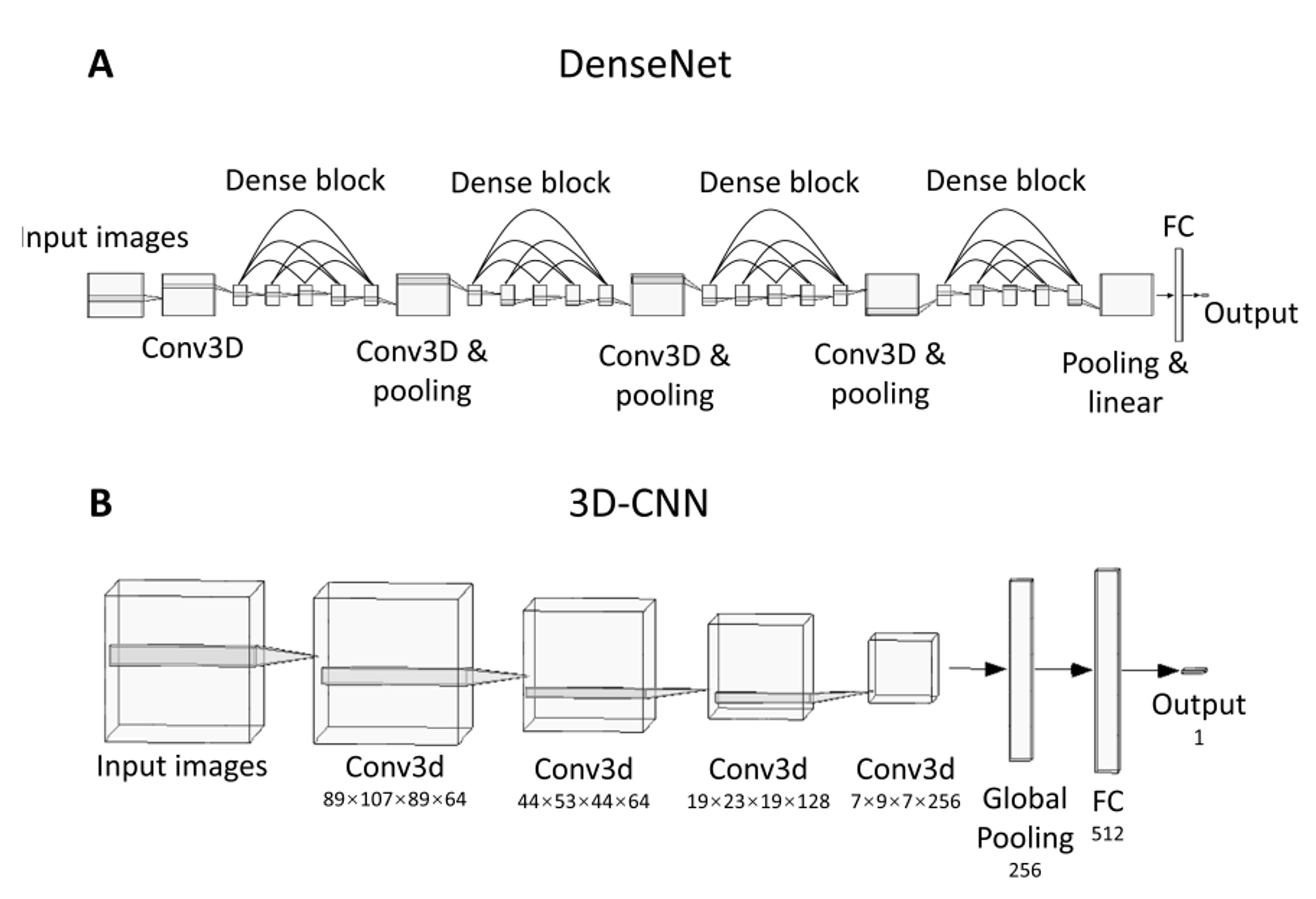
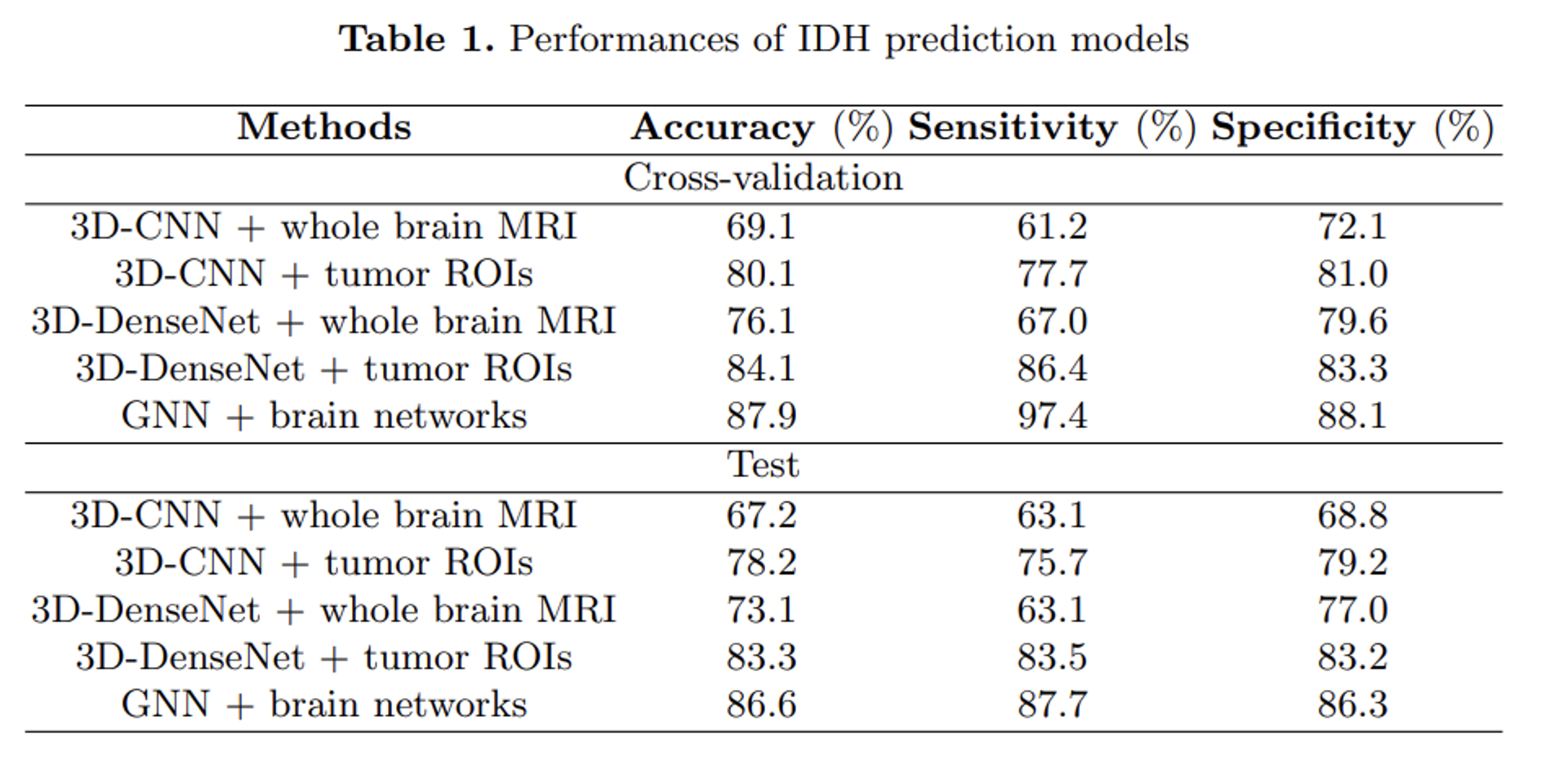
- Tumor voxel을 입력으로 받는 비교 벤치마크 네트워크(3DCNN, DenseNet)의 성능이 전체 MRI를 받은 성능보다 높게 나왔으며, 이를 통해 local region을 넘어서 확장된 뇌 네트워크의 잠재적인 bias가 학습 과정에서 활용되고 있음을 확인해볼 수 있습니다.
- Atlas 및 뇌 네트워크를 통합적으로 활용한 제안 모델은 비교 벤치마크 성능보다 더 좋았으며, 이로부터 뇌 네트워크에 임베딩되어진 사전 지식정보의 통합이 그래프 모델의 local 및 global한 종양 침투 부분들 사이의 유의미한 특징정보를 학습하고 추출할 수 있었음을 보여주고 있습니다.
- 추가적으로 GNN 모델의 학습과정을 해석하기 위해, GNNExplainer를 적용하여 엣지 중요도 및 하위 네트워크 출력을 시각화하여 아래와 같이 제공합니다.
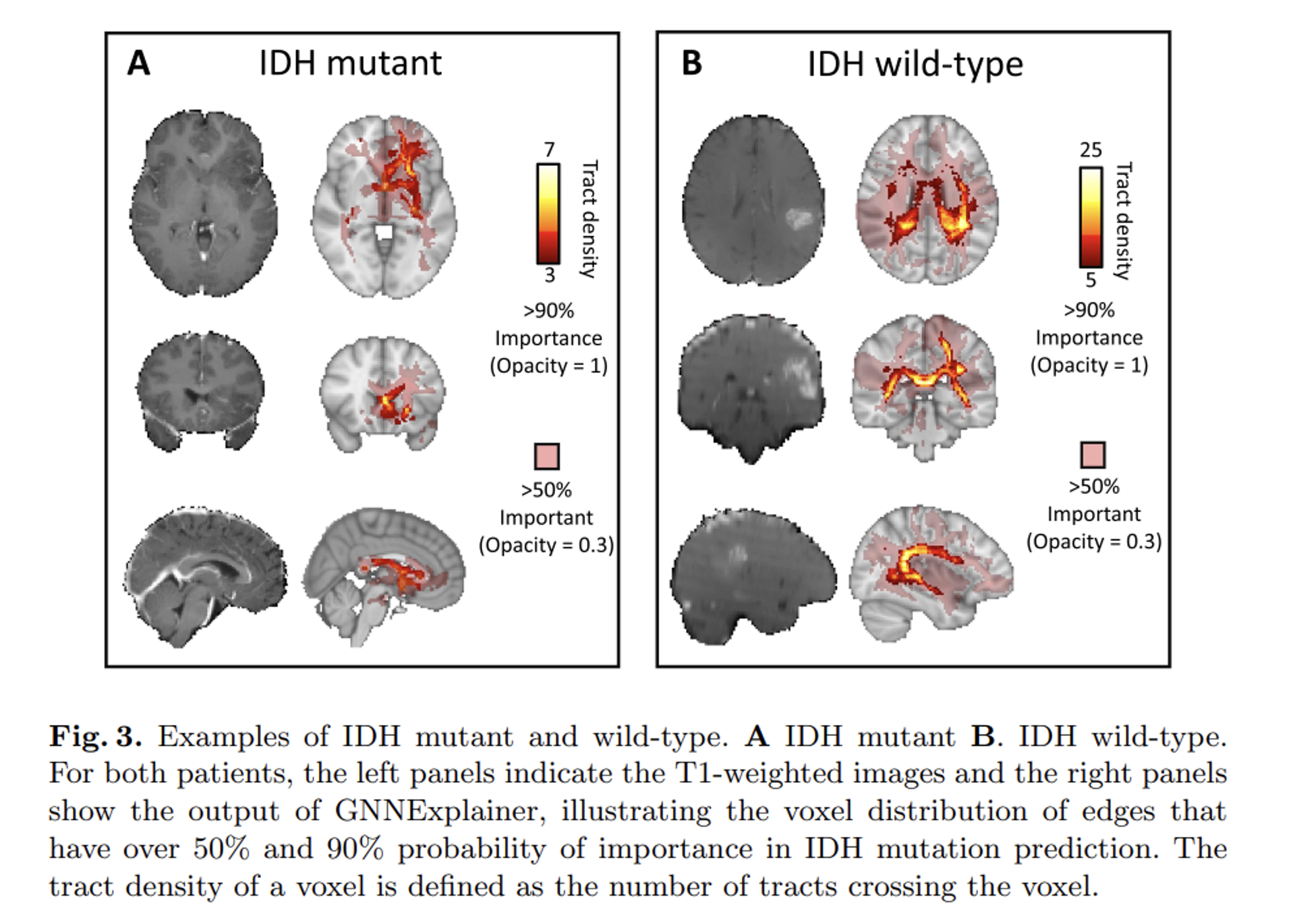
- 전체적으로 wild-type에서 GNN 모델이 포착해낸 더 넓은 엣지 상의 종양 분포도를 효과적으로 확인해볼 수 있으며, 그로부터 IDH mutant Glioma의 광범위한 뇌 네트워크 침입 tract 정보를 연관지어 포착할 수 있는 GNN 방식의 장점을 확인해볼 수 있습니다.
- 핵심 포인트를 간략히 잡아보자면, 복합적 요소들을 고려해야 하는 Complex network로 정의되는 뇌 네트워크를 충분히 그래프 특징들을 활용하여 커버할 수 있습니다. 또한 기존 방법들로는 고려하기 어려운 매우 복잡한 정보 (종양의 침투 경로 및 분포도)에 대한 사전정보와의 관계형 학습이 가능한 GNN의 메커니즘적 장점 등을 충분히 확인해볼 수 있었던 것 같습니다.
[Contact Info]
Gmail : jhbae1184@akane.waseda.jp
Twitter (X): @jhbae1184
